분량+저녁크리로 인해 2부작으로 나눠버림...
이 글의 re모듈은 파이썬에서 사용하는거지만, 앞 글에 있는 기호와 메타문자는 언어를 불문하고 정규 표현식에서 다 쓰인다.
https://koreanraichu.tistory.com/118
정규식(Regular Expression)-기호와 메타문자
문자 찾는 것 자체는 find()도 해주는데, 얘는 딱 정확하게 일치하는 문자열만 찾아준다. 그럼 정규식은? 그건 대충 와일드카드같은 거다. 그러니까 find()는 소라빵 찐빵 팥빵 붕
koreanraichu.tistory.com
다른 언어를 사용한다고 해도 앞 글은 한번정도는 읽어보자.
re.search()
자... 전에도 서술했지만, find()는 정규식을 주면 그 정규식이랑 일치하는 걸 찾지 정규식으로 검색하질 않아요...
print(re.search('G...',DNA))<re.Match object; span=(1, 5), match='GAGG'>단식
for i in range(len(df)):
enzyme = df['Enzyme'][i]
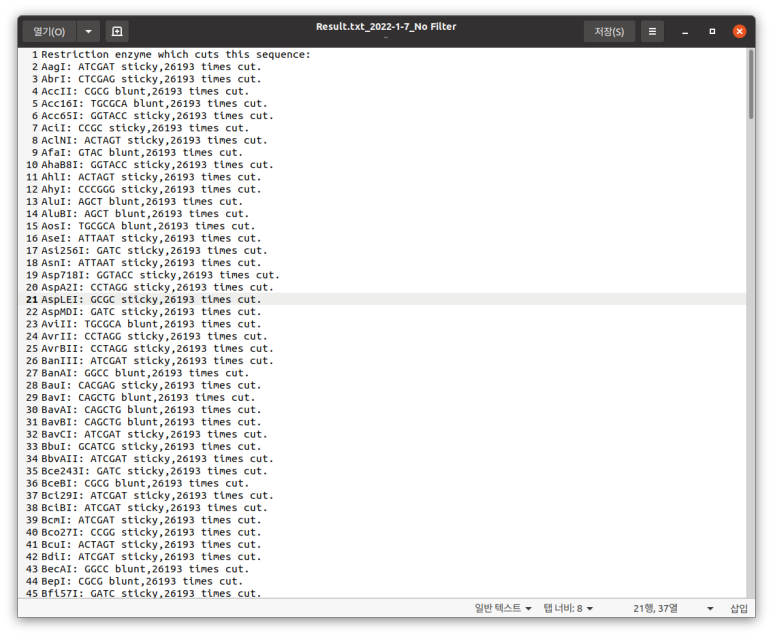
if re.search('A.....',enzyme):
print(enzyme)Acc36I
AccB1I
AccB2I
AccB7I
AceIII
AflIII
Alw21I
Alw26I
Ama87I
AmaCSI
ApaORI
AquIII
Asp10HII
Asp26HI
Asp27HI
Asp35HI
Asp36HI
Asp40HI
Asp50HI
Asp700I
Asp745I
Asp59I
AsuIII
AsuC2I
AsuHPI복식
일단 자세한건 re.match()에서 서술하겠지만, 일단 re.search()가 정규식 검색할 때 쓰는 게 맞다.
re.match()
print(re.match('G...',DNA))Nonere.search()와 달리 re.match()는 처음부터 정규식 형식이랑 일치하는 걸 찾아준다. 쟤가 그래서 왜 None이냐...
DNA="AGAGGTTAAAGAACAAAGGCTTACTGTGCGCAGAGGAACGCCCATTTAGCGGCTGGCGTTTTGAATCCTCGGTCCCCCTTGTCTATCCAGATTAATCCAATTCCCTCATTTAGGACCCTACCAAGTCAACATTGGTATATGAATGCGACCTCGAAGAGGCCGCCTAAAAATGACAGTGGTTGGTGCTCTAAACTTCATTTGGTTAACTCGTGTATCAGCGCGATAGGCTGTTAGAGGTTTAATATTGTATGGCAAGGTACTTCCGGTCTTAATGAATGGCCGGGAAAGGTACGCACGCGGTATGGGGGGGTGAAGGGGCGAATAGACAGGCTCCCCTCTCACTCGCTAGGAGGCAATTGTATAAGAATGCATACTGCATCGATACATAAAACGTCTCCATCGCTTGCCCAAGTTGTGAAGTGTCTATCACCCCTAGGCCCGTTTCCCGCATATTAACGCCTGATTGTATCCGCATTTGATGCTACCGTGGTTGAGTCAGCGTCGAGCACGCGGCACTTATTGCATGAGTAGAGTTGACTAAGAGCCGTTAGATGCCTCGCTGTACTAATAGTTGTCGACAGATCGTCAAGATTAGAAAACGGTAGCAGCATTATCGGAGGTTCTCTAACTAGTATGGATAGCCGTGTCTTCACTGTGCTGCGGCTACCCATCGCCTGAAAACCAGTTGGTGTTAAGCGATCCCCTGTCCAGGACGCCACACGTAGTGAAACATACACGTTCGTCGGGTTCACCCGGGTCGGATCTGAGTCGACCAAGGACACACTCGAGCTCCGATCCCTACTGTCGAGAAATTTGTATCCCGCCCCCGCAGCTTGCCAGCTCTTTGAGTATCATGGAGCCCATGGTTGAATGAGTCCAATAACGAACTTCGACATGATAAAGTCCCCCCCTCGCGACTTCCAGAGAAGAAGACTACTGAGTTGAGCGTTCCCAGCACTTCAGCCAAGGAAGTTACCAATTTTTAGTTTCCGAGTGACAC"원본 시퀀스가 이거였음. G로 시작하는 게 첫 번째 글자가 아니라 두 번째 글자기때문에 첫 빠따부터 에이 뭐야 없어가 되버린다.
print(re.match('.G...',DNA))
print(re.search('.G...',DNA))<re.Match object; span=(0, 5), match='AGAGG'>
<re.Match object; span=(0, 5), match='AGAGG'>이거는 둘 다 똑같은 결과가 나온다. (정규식 형식은 어쨌든 일치하므로)
re,match()의 메소드들
이거 참고로 단독소환 쌉가능.
records = re.finditer('A.{4}C',DNA)
for r in records:
print(r.group(),r.start(),r.end(),r.span())finditer에 대해서는 밑에서 또 설명할 예정이니까 이것부터 보고 갑시다. 저기 r.뭐시기 된 저거요. 저게 re.match의 메소드인데 순서대로
1) 그룹(문자열)
2) 시작
3) 끝
4) span
이렇게 된다. 출력은
AAGAAC 8 14 (8, 14)이렇게 된다.
re.findall()
이름을 보면 아시겠지만 그냥 다 찾아주는 애다. 반복문 데려 올 필요도 없다.
print(re.findall('G...',DNA))['GAGG', 'GAAC', 'GGCT', 'GTGC', 'GCAG', 'GGAA', 'GCCC', 'GCGG', 'GGCG', 'GAAT', 'GGTC', 'GTCT', 'GATT', 'GGAC', 'GTCA', 'GGTA', 'GAAT', 'GCGA', 'GAAG', 'GGCC', 'GCCT', 'GACA', 'GTGG', 'GGTG', 'GGTT', 'GTGT', 'GCGC', 'GATA', 'GGCT', 'GTTA', 'GAGG', 'GTAT', 'GGCA', 'GGTA', 'GGTC', 'GAAT', 'GGCC', 'GGGA', 'GGTA', 'GCAC', 'GCGG', 'GGGG', 'GGGT', 'GAAG', 'GGGC', 'GAAT', 'GACA', 'GGCT', 'GCTA', 'GGAG', 'GCAA', 'GTAT', 'GAAT', 'GCAT', 'GCAT', 'GATA', 'GTCT', 'GCTT', 'GCCC', 'GTTG', 'GAAG', 'GTCT', 'GGCC', 'GTTT', 'GCAT', 'GCCT', 'GATT', 'GTAT', 'GCAT', 'GATG', 'GTGG', 'GAGT', 'GCGT', 'GAGC', 'GCGG', 'GCAT', 'GAGT', 'GAGT', 'GACT', 'GAGC', 'GTTA', 'GATG', 'GCTG', 'GTTG', 'GACA', 'GATC', 'GTCA', 'GATT', 'GAAA', 'GGTA', 'GCAG', 'GGAG', 'GTTC', 'GTAT', 'GGAT', 'GCCG', 'GTCT', 'GTGC', 'GCGG', 'GCCT', 'GAAA', 'GTTG', 'GTGT', 'GCGA', 'GTCC', 'GGAC', 'GCCA', 'GTAG', 'GAAA', 'GTTC', 'GTCG', 'GGTT', 'GGGT', 'GGAT', 'GAGT', 'GACC', 'GGAC', 'GAGC', 'GATC', 'GTCG', 'GAAA', 'GTAT', 'GCCC', 'GCAG', 'GCCA', 'GCTC', 'GAGT', 'GGAG', 'GGTT', 'GAAT', 'GAGT', 'GAAC', 'GACA', 'GATA', 'GTCC', 'GCGA', 'GAGA', 'GAAG', 'GAGT', 'GAGC', 'GTTC', 'GCAC', 'GCCA', 'GGAA', 'GTTA', 'GTTT', 'GAGT', 'GACA']근데 위치까지 준다고는 안 했다.
re.finditer()
print(re.finditer('A..C',DNA))<callable_iterator object at 0x7f009c5f8100>얘는 다른 메소드들처럼 걍 쌩으로 print문에 넣으면 안된다. 저렇게 뜬다.
records = re.finditer('A..C',DNA)
for r in records:
print(r)<re.Match object; span=(16, 20), match='AGGC'>
<re.Match object; span=(37, 41), match='ACGC'>
<re.Match object; span=(63, 67), match='AATC'>
<re.Match object; span=(84, 88), match='ATCC'>
<re.Match object; span=(93, 97), match='AATC'>
<re.Match object; span=(99, 103), match='ATTC'>
<re.Match object; span=(114, 118), match='ACCC'>
<re.Match object; span=(123, 127), match='AGTC'>
<re.Match object; span=(142, 146), match='ATGC'>
<re.Match object; span=(156, 160), match='AGGC'>
<re.Match object; span=(189, 193), match='AAAC'>
<re.Match object; span=(205, 209), match='ACTC'>
<re.Match object; span=(224, 228), match='AGGC'>
<re.Match object; span=(290, 294), match='ACGC'>
<re.Match object; span=(294, 298), match='ACGC'>
<re.Match object; span=(323, 327), match='AGAC'>
<re.Match object; span=(327, 331), match='AGGC'>
<re.Match object; span=(340, 344), match='ACTC'>
<re.Match object; span=(350, 354), match='AGGC'>
<re.Match object; span=(366, 370), match='ATGC'>
<re.Match object; span=(370, 374), match='ATAC'>
<re.Match object; span=(381, 385), match='ATAC'>
<re.Match object; span=(388, 392), match='AAAC'>
<re.Match object; span=(428, 432), match='ACCC'>
<re.Match object; span=(434, 438), match='AGGC'>
<re.Match object; span=(455, 459), match='ACGC'>
<re.Match object; span=(467, 471), match='ATCC'>
<re.Match object; span=(478, 482), match='ATGC'>
<re.Match object; span=(493, 497), match='AGTC'>
<re.Match object; span=(507, 511), match='ACGC'>
<re.Match object; span=(542, 546), match='AGCC'>
<re.Match object; span=(551, 555), match='ATGC'>
<re.Match object; span=(596, 600), match='AAAC'>
<re.Match object; span=(639, 643), match='AGCC'>
<re.Match object; span=(665, 669), match='ACCC'>
<re.Match object; span=(678, 682), match='AAAC'>
<re.Match object; span=(693, 697), match='AAGC'>
<re.Match object; span=(698, 702), match='ATCC'>
<re.Match object; span=(712, 716), match='ACGC'>
<re.Match object; span=(717, 721), match='ACAC'>
<re.Match object; span=(727, 731), match='AAAC'>
<re.Match object; span=(731, 735), match='ATAC'>
<re.Match object; span=(750, 754), match='ACCC'>
<re.Match object; span=(766, 770), match='AGTC'>
<re.Match object; span=(778, 782), match='ACAC'>
<re.Match object; span=(782, 786), match='ACTC'>
<re.Match object; span=(794, 798), match='ATCC'>
<re.Match object; span=(817, 821), match='ATCC'>
<re.Match object; span=(857, 861), match='AGCC'>
<re.Match object; span=(873, 877), match='AGTC'>
<re.Match object; span=(901, 905), match='AGTC'>
<re.Match object; span=(930, 934), match='AGAC'>
<re.Match object; span=(961, 965), match='AGCC'>
<re.Match object; span=(996, 1000), match='ACAC'>Biopython에서 parse로 불러오는것마냥 for문이 필요하다.
전방탐색
text='http://localhost:8888/notebooks/re.search.ipynb'이런 텍스트가 있을 때
p = re.compile(".+:")
print(p.search(text))보통은 이렇게 찾는다. 근데 이 방식에는 문제가 있어요... 정규식이 복잡해지면 정규식 짜다가

이러고 밥상 엎는다.
긍정형 전방 탐색
p = re.compile(".+(?=:)")
print(p.search(text))<re.Match object; span=(0, 16), match='http://localhost'>?=가 들어가는 것. 저 식의 경우 :가 있으면 응 있네 여이따 하고 찾아는 주지만 출력을 안 한다. (저거는 Jupyter URL로 해서 그렇다)
부정형 전방 탐색
여러분들은 불신의 아이콘에 대해 아시는가?

얘 맞다.

그러니까 이런 거.
블로그에서 뭔가를 검색했는 데 저게 보이면 일단 거르고 보잖음. 근데 쟤랑 그거랑 뭔 상관이냐고? 자, 생각해봅시다... 일반적으로 맛집을 찾을 때 저런 바이럴들이 많이 나오니까 우리는 저것들을 피하기 위한 키워드를 정해서 맛집을 찾잖음? (오빠랑 맛집 뭐 이런걸로다가) 부정형 전방 탐색은 맛집 검색을 하면서 아예 저 불신의 아이콘을 빼버리고 찾는 거라고 보면 됨.
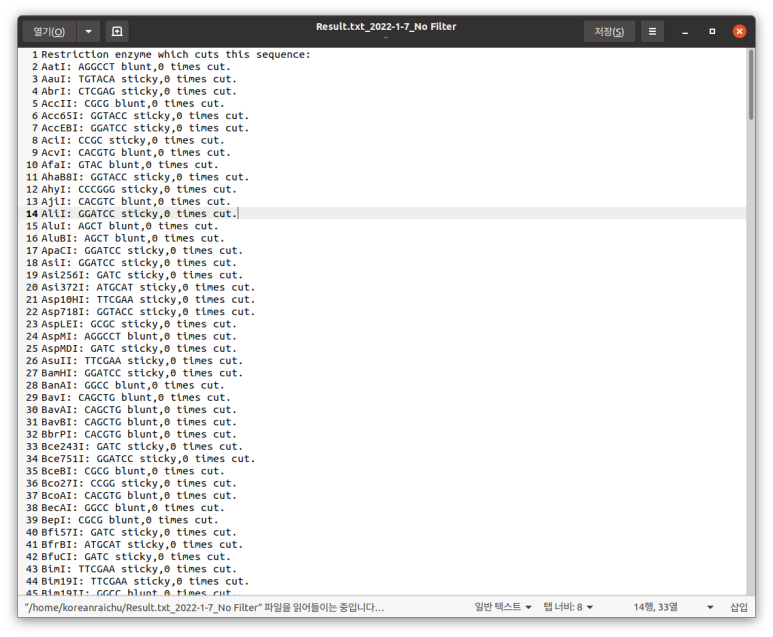
file_list=['buchu.jpg','moon.jpg','text.txt','py.py','BOJ.ipynb','jemok.png','pyo.csv']그러니까 이런 파일들이 있을 때 jpg파일을 빼려면 txt, py, ipynb, png, csv파일을 묶어야 하는데 정규식에 어느 세월에 저걸 다 쓰고 앉았음?
for i in file_list:
if re.match('.*[.](?!jpg$).*$',i):
print(i)text.txt
py.py
BOJ.ipynb
jemok.png
pyo.csv이럴 때 부정형 전방탐색으로 jpg를 빼버리면 된다. 정확히는 jpg가 포함되지 않은 것만 보여준다.
sub(), subn()
p=re.compile('W')
p.sub('[A|T]','CCWGG')이런 식으로 쓰면 W가 A or T로 바뀌어서 나온다.
p=re.compile('N')
p.sub('.','GATNNNATCNNNNNNNN',count=3)이런 식으로 바꿀 횟수를 지정하는 것도 가능하다. (count=n)
('GAT...ATC........', 11)subn()도 쓰는 방법은 비슷한데, 얘는 반환 형태가 튜플이고 몇 글자 바꿨는지가 나온다.
re.compile()
정규식 일일이 쓰기 귀찮을 때 쓰면 좋다.
p = re.compile('G....C')
print(p.search(DNA))compile에 옵션을 지정하고, 그 다음 match나 search, 파인드올 돌리면 된다.
p = re.compile('t.....t',re.I)
print(p.findall(DNA))옵션은 이런 식으로 붙고, 총 네 개가 있다.
re.compile()의 옵션
1)Dotall(S):와일드카드를 ㄹㅇ 와일드카드로 쓸 수 있다. 저 옵션이 없으면 개행문자는 와일드카드로 대체가 안되는데, 저 옵션을 붙여벼리면 아 그런건 모르겠고 와일드카드! 가 된다.
2) Ignorecase(I): 대소문자를 무시하고 그냥 일치하는 문자열을 찾아준다. 그러니까 저 옵션을 주면 A랑 a랑 같아진다. (원래 파이썬은 대소문자 칼같이 구별한다)
3) Multiline(M): 여러 줄로 된 문자열을 줄단위로 인식한다. 그러니까 이게 무슨 말이냐...
나 보기가 역겨워
가실 때에는
말없이 고이 보내 드리오리다
영변에 약산
진달래꽃
아름 따다 가실 길에 뿌리오리다
가시는 걸음걸음
놓인 그 꽃을
사뿐히 즈려밟고 가시옵소서
나 보기가 역겨워
가실 때에는
죽어도 아니 눈물 흘리오리다이 시는 김소월의 진달래꽃이다. 마야 아니다 우리가 볼 때는 4연이고 각 연마다 3행인데, Multiline이 없는 정규식 처리에서는 저걸 그냥 한 줄로 본다. 행이고 연이고 그런건 모르겠고 걍 한줄이여! 이렇게 되버리는데 Multiline 옵션이 들어가면 12줄짜리 텍스트(연과 연 사이 개행문자 포함 15줄)로 본다.
4) Verbose(X): 정규식에 주석을 넣을 수 있다. 코드와 마찬가지로 정규식도 복잡해지면

이렇게 된다. (아니면 머리에 블루스크린이 뜨거나) Verbose 옵션을 주면 그걸 방지하고자 주서을 넣을 수 있게 된다.
'Coding > Python' 카테고리의 다른 글
| Cutter, Finder 공통 패치: FASTA 파일 불러오는 기능 (0) | 2022.08.22 |
|---|---|
| Finder 기능추가: 정규식 도입 (0) | 2022.08.22 |
| 정규식(Regular Expression)-기호와 메타문자 (0) | 2022.08.22 |
| Cutter 기능 추가: 정규식 도입 (0) | 2022.08.22 |
| Searcher 기능 추가: 그 뭐더라 그 D로 시작하는 그거 (0) | 2022.08.21 |