근데 NEB커터 쓰세여 그거 좋음
일단 이 코드는 이 시퀀스를 자르는 제한효소들을 찾는 게 아님. 이 효소가 이 시퀀스를 자르는가? 를 보는 코드임다. 이 점 유념해주세요. 그리고 이거 올리면서 Jupyter가 매트랩이랑 비중 같아졌다
코드
Jupyter notebook으로 코딩한거고 나중에 일부 블록은 정리할 예정.
import pandas as pd구축한 csv파일을 가져오고 취급하려면 얘가 필요하다. csv파일은 혹시 써보실 분 계시면 말씀주세요.
참고로 csv파일이 되게 단촐해서 효소 이름, 인식하는 시퀀스, 자르는 시퀀스, 자르는 형태(sticky or blunt)가 들어가 있음. 나중에 여건이 된다면 똑같은 부분을 인식하고 자르는 다른 효소나 처리온도에 대한 정보도 추가할 예정. (물론 출력 파일에도 같이 나오게 해야 하는 게 함정)
enzyme_table = pd.read_csv('/home/koreanraichu/restriction.csv') # 일단 자체적으로 구축했음... 저거 종류 개많습니다 ㅠㅠ
enzyme_table = enzyme_table.sort_values('Enzyme')
# csv파일의 구성은 크게 효소 이름, 인식하는 시퀀스, 해당 시퀀스를 자르는 형태와 sticky or blunt 여부로 구성되어 있습니다.위에서 얘기한 csv파일을 불러와서 효소 이름순으로 정렬합니다. 참고로 효소가 인식하는 시퀀스에 N, W, D, S같은 거(ATGC 말고 다른 거) 들어가 있는 효소는 일단 뺐음. 걔들은 따로 처리해야 하는데 그것까지는 아직 무리입니다.
enzyme=input('시퀀스를 찾을 제한효소를 입력해주세요: ')
search_sequence=input('제한효소 site를 찾을 시퀀스를 입력해주세요: ')이거는 입력받는 코드. 효소 이름은 토씨 하나 안 틀리고 써야 한다.
if enzyme_table['Enzyme'].isin([enzyme]).any() == True:
res_find = enzyme_table.sequence[(enzyme_table['Enzyme'] == enzyme)]
res_find = res_find.to_string(index=False)
res_find = str(res_find)
print(res_find)
# 효소 이름이 데이터베이스에 있을 경우 검색할 시퀀스 데이터를 가져온다
res_site = enzyme_table.restriction_site[(enzyme_table['Enzyme'] == enzyme)]
res_site = res_site.to_string(index=False)
res_site = str(res_site)
print(res_site)
# 효소 이름이 데이터베이스에 있을 경우 검색하고 대체할 시퀀스 데이터를 가져온다
cut_feature = enzyme_table.cut_feature[(enzyme_table['Enzyme'] == enzyme)]
cut_feature = cut_feature.to_string(index=False)
cut_feature = str(cut_feature)
print(cut_feature)
# blunt or sticky(나중에 저장 기능 추가할 때 넣을 예정입니다)
else:
print("No data in Database")판다스 종특인지는 모르겠으나... to_string() 없으니까 인식 못하데...
순서대로 '인식하는 시퀀스', '자르는 시퀀스', '자르는 형태' 컬럼. 이 코드 자체는 가져온 DB에 효소 이름이 있어야 진행하므로 DB에 효소가 없을 때는 No data가 뜬다. (참고로 수동으로 구축중이라 시간 무지하게 걸림)
if enzyme_table['Enzyme'].isin([enzyme]).any() == True:
print(search_sequence.find(str(res_find)))
else:
pass
# 여기는 검색결과가 존재하지 않으면 -1로 나옵니다. (윗 블럭이랑 여기는 넘어가도 되는 부분)이 코드도 마찬가지로 DB에 효소가 있을 때 시퀀스를 찾고, 결과를 출력한다. (find.()는 문자열이 없으면 -1로 뽑음)
with open ('Result.txt','w',encoding='utf-8') as f:
if search_sequence.find(res_find) != -1:
search_sequence = search_sequence.replace(res_find,res_site)
print(enzyme,",",cut_feature)
print(search_sequence)
f.write(enzyme)
f.write(", ")
f.write(res_site)
f.write(", ")
f.write(cut_feature)
f.write("\n")
f.write(search_sequence)
f.close()
# DB에 효소가 있고 일치하는 시퀀스가 있을 때
elif enzyme_table['Enzyme'].isin([enzyme]).any() == True and search_sequence.find(res_find) == -1:
print("No restriction site in this sequence. ")
f.write(enzyme)
f.write(", ")
f.write(res_site)
f.write(", ")
f.write(cut_feature)
f.write("\n")
f.write("This restricion enzyme never cut this sequence. ")
f.close()
# DB에 효소가 있으나 일치하는 시퀀스가 없을 때
else:
print("No data in database. ")
f.write(enzyme)
f.write("\n")
f.write("This restriction enzyme not entried in database. ")
f.close()
# DB에 효소가 없을 때출력과 저장에 대한 코드. 크게 DB에 효소가 있나/없나로 갈리고 효소가 있을 때 인식 시퀀스가 있나/없나로 갈린다.
그래서 각각
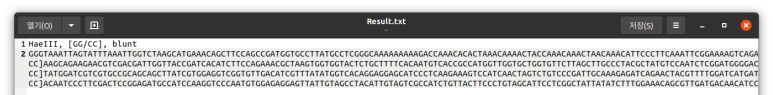


이런 식으로 출력된다.
앞으로 추가할 기능
1. csv 파일 수정에 따른 출력 형식(현재 효소 이름, 자르는 시퀀스, sticky ot blunt 여부만 있는데 처리 온도도 및 같은 시퀀스를 인식하는 다른 효소의 정보도 추가할 예정)
2. 출력 파일명도 형식을 줄 수 있으면 날짜 이런거 들어가면 좋을 것 같음. (아니면 사용자가 입력할 수 있게 하거나) 일단 오늘 날짜 가져오는 게 먼저긴 한데 아니면 하이브리드로 입력 받아서 오늘 날짜랑 같이 묶는 뭐 이런거?
멀티랑 N, W, K 처리는... 아 그건 제 능력 밖이니까 걍 NEB 커터 쓰세여... 그거 좋음...
'Coding > Python' 카테고리의 다른 글
| 제한효소 커터 2편 나왔음 (0) | 2022.08.21 |
|---|---|
| 제한효소 커터 코드 패치했음 (0) | 2022.08.21 |
| 오케이 따옴표 떼버렸음 (0) | 2022.08.21 |
| 10진수->2진수 변환 코드 (0) | 2022.08.21 |
| 번외편-코딩테스트 풀이 (3) (0) | 2022.08.21 |

